医療・健康
見逃されていた細胞ごとのばらつきを可視化するソフトウェアを開発
筑波大学 医学医療系・人工知能科学センターの尾崎遼准教授、国立研究開発法人理化学研究所 生命機能科学研究センターの二階堂愛チームリーダーらの研究グループは、1細胞RNAシーケンス法(1細胞RNA-seq)データのリードカバレッジの細胞間変動(細胞ごとのばらつき)を可視化するソフトウェア「Millefy(ミルフィー)」を開発しました。
1細胞RNA-seqデータのリードカバレッジには、既知・新規転写単位、正常・異常スプライシング、アンチセンスRNAやエンハンサーRNAの発現などの様々なRNA関連生命現象が反映されています。しかし、これを効果的に捉えるデータ可視化手法はこれまで存在しませんでした。
本研究では、リードカバレッジの細胞間変動を可視化するソフトウェア「Millefy(ミルフィー)」を開発しました。Millefyを、既報のマウス胚性幹細胞(ES細胞)およびトリプルネガティブ乳がん患者由来上皮細胞の1細胞RNA-seqデータに適用したところ、アンチセンスRNAの制御、エンハンサーRNAの発現、非翻訳領域長といった、遺伝子発現量だけでは発見・解釈が困難な細胞間変動を明らかにできることがわかりました。
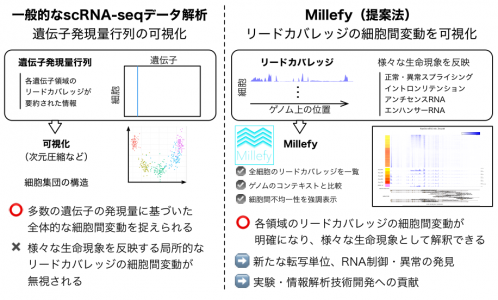
図 本研究の概要
1細胞RNA-seqデータのリードカバレッジには、既知・新規転写単位、正常・異常スプライシング、アンチセンスRNAやエンハンサーRNAの発現などの様々なRNA関連生命現象が反映されています。しかし、これを効果的に捉えるデータ可視化手法はこれまで存在しませんでした。
本研究では、リードカバレッジの細胞間変動を可視化するソフトウェア「Millefy(ミルフィー)」を開発しました。Millefyを、既報のマウス胚性幹細胞(ES細胞)およびトリプルネガティブ乳がん患者由来上皮細胞の1細胞RNA-seqデータに適用したところ、アンチセンスRNAの制御、エンハンサーRNAの発現、非翻訳領域長といった、遺伝子発現量だけでは発見・解釈が困難な細胞間変動を明らかにできることがわかりました。

図 本研究の概要
PDF資料
プレスリリースPREVIOUS
医療・健康
T細胞リンパ腫に他の血液がんの薬剤が有望であることを発見
